|

|

|

|

|

|

|
|
|
|
Infos
/ Organisation Meeting
|
 
English
Version available
|
Reférences
Bibliographiques / References
|
Infos
Séquençages / Sequencing info
|
Contact
Participants / Reach Attendies
|
Crédits
/ Credits
|
|
|
|
Pour la
première fois, des scientifiques venant d'horizons
différents se sont réunis pour
réfléchir sur la nature du dernier
ancêtre commun à tous les êtres vivants
actuels. La fondation des Treilles a permis l'organisation
sur ce thème d'un colloque sortant du cadre habituel
des conférences scientifiques. De nombreux
participants ont présenté plusieurs
communications, certaines d'entre elles improvisées.
Très vite, l'atmosphère de convivialité
propre aux Treilles a fait naître chez tous les
membres de notre petit groupe (dont beaucoup ne
s'étaient jamais rencontrés auparavant) le
sentiment d'appartenir à une même
communauté, à l'aube d'une nouvelle aventure
scientifique. Dans un domaine (les premiers pas de la vie
sur terre) où les confrontations d'idées (et
parfois de personnalités) peuvent être vives,
l'atmosphère lumineuse de la Provence permettait la
discussion des hypothèses les plus contradictoires en
toute sérénité.
|
|
|
|

|
L'objectif
premier du colloque était de rassembler des
chercheurs appartenant à des "tribus" scientifiques
différentes. Chaque tribu a ses rites propres, en
particulier ses grandes conférences organisées
à intervalles réguliers, ses journaux de
prédilection et ses grands ténors. Les membres
de ces différents clans se mélangent rarement,
et seuls quelques audacieux vont parfois jusqu'à se
faire accepter dans deux ou trois tribus différentes.
Aux Treilles, cinq d'entre elles au moins étaient
représentées : celle des chercheurs
passionés par le problème des origines de la
vie, dont le grand
meeting international, qui a lieu tous les trois ans,
venait juste de se tenir à Orléans, celle des
archaeo-microbiologistes, spécialistes du
troisième groupe d'êtres vivants sur terre, les
archaeabactéries ou archaées, celle des
thermophilistes, qui étudient la vie microbienne
à très haute température
(jusqu'à 110°C), celle des évolutionistes
moléculaires, qui essayent de retrouver la
généalogie de tous les êtres vivants
(l'arbre universel) en comparant les séquences de
leurs macromolécules, et enfin celle des
génomistes, une nouvelle tribu en formation, dont
l'objectif est l'analyse exhaustive des génomes,
rendue possible grâce aux grands programmes de
séquençage systématique de l'ADN.
|
|
Quelques
participants n'appartenaient à aucune des cinq tribus
mentionnées, ils avaient été choisis en
raison de leur compétence reconnue dans un domaine
dont l'importance paraissait cruciale pour aborder le
problème du dernier ancêtre universel. Certains
étaient confrontés pour la première
fois aux grandes questions posées par
l'évolution du vivant, et ils ne furent pas les moins
enthousiastes.
|
|
|
|
|
La
plupart des chercheurs présents étaient
des expérimentalistes, ils ont derrière eux
une longue tradition de prudence, de rigueur, allant parfois
jusqu'à la méfiance pour toute idée
trop spéculative. En particulier, le
réductionisme de la biologie moléculaire et la
coupure qui s'est créée depuis cinquante ans
entre les biochimistes "purs et durs" et les
évolutionistes sont des obstacles majeurs dans un
domaine où l'imagination joue un rôle
déterminant. De ce point de vue,
Christian de Duve a joué
un rôle irremplaçable au cours de ces
journées. Prix Nobel, doyen de notre colloque, et
reconnu pour ses travaux de biologie cellulaire, il a fait
preuve d'une imagination fertile, jouant un rôle
particulièrement actif dans nos discussions. Nous
espérons tous maintenant que le colloque des Treilles
aura été l'acte fondateur d'une nouvelle
thématique de recherche à part entière
: à la recherche de LUCA
(the Last Universal Common Ancestor).
|
|
LUCA
a été baptisé aux Treilles, nous ne
savons pas encore si ce nom s'imposera, il ne fait
même pas l'unanimité entre nous. D'autres
termes ont été proposés au cours des
années : le
nom progenote, avancé en 1977 par
Carl Woese (le créateur
du concept d'archaebactéries) a eu son heure de
gloire, mais il semble sur le déclin. La notion de
progénote est en effet associée à la
conception d'un ancêtre particulièrement
primitif, beaucoup plus simple que la plus simple des
cellules actuelles, ce qui ne correspond plus à la
pensée de nombreux spécialistes. La vision
majoritaire aujourd'hui est plutôt celle d'un
ancêtre commun ressemblant aux bactéries ou aux
archaées, et pour quelque uns, celle d'une
créature intermédiaire entre les procaryotes
(cellules sans noyau) et les eucaryotes (cellules avec
noyau).
|

|
|
|
Le
terme cenancestor
(de la racine grec cen, ensemble) proposé par
Fitch en 1987, a la faveur
des puristes. Pour d'autres, il présente
l'inconvénient d'être incompréhensible
au commun des mortels, et même au commun des
biologistes. L'appellation "dernier ancêtre commun"
(Last Common Ancestor) est de plus en plus utilisée
dans la littérature.
José Castresana, lors de
notre colloque a fait remarquer qu'il est aussi trop vague.
Le terme "dernier ancêtre commun" peut être
utilisé (et il l'est en effet) pour tout groupe
d'organismes.
|
|
|
|
LUCA est
un compromis entre LCA et LUA (last universal ancestor)
proposé à ce colloque par
Christos Ouzounis. Il pourrait
être médiatique, de
LUCA à LUCY
résumant la trajectoire de l'évolution du
vivant. Il définit une entité sympathique,
c'est lui qui a ensemencé notre planète, nous
sommes tous ses descendants.
|
|
Pourquoi
focaliser notre attention sur ce personnage clef de notre
histoire ? Pourquoi un colloque sur ce thème
aujourd'hui ? Pendant longtemps la recherche sur les
origines de la vie s'est uniquement consacrée
à comprendre comment étaient apparues les
premières molécules du vivant sur notre
planète. Une fois celles-ci présentes, la
suite allait de soi. Il n'y avait pas de place pour
LUCA dans ces recherches. De
leur côté, les biologistes cellulaires et
moléculaires (à de très rares
exceptions) n'allaient pas perdre leur temps (très
précieux) à spéculer sur un
ancêtre insaisissable, alors que s'offrait à
eux l'inépuisable richesse de modèles
d'études, bien réels ceux-là (en
protéines et en acides nucléiques).
|
|
|
|
La
situation a bien changé aujourd'hui,
précisément grâce aux progrès de
la biologie moléculaire. On s'est aperçu, en
comparant les molécules du vivant chez tous les
organismes (des bactéries à l'homme) qu'elles
présentaient des points communs, mémoire
fossilisée de leur lointain ancêtre. Ce sont
ces recherches qui ont conduit à découvrir la
présence sur terre d'un groupe de procaryotes, les
archaées, aussi éloignés des
bactéries que des eucaryotes.
|
|
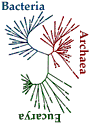
En
comparant les mécanismes fondamentaux du
vivant dans les trois domaines (bactéries,
archaées et eucaryotes) il devenait alors possible de
définir "le plus petit dénominateur commun aux
trois" : LUCA.
|
|
|
|
|
|
Il
restait à faire le lien entre les biologistes
amoureux de l'évolution, qui se rapprochent ainsi des
origines premières en remontant dans le passé,
et les pionniers des recherches sur les origines de la vie,
qui essaient depuis l'expérience
fondatrice de Stanley
Miller en 1953 de partir du point 0 (soupe primitive
ou aurore de pierre) pour avancer vers le présent. Le
temps était venu de rassembler les tribus.
|

|
|
Quelques questions débattues aux
Treilles
|
|
Est-il possible de dresser un
portrait-robot de
LUCA
?
|
|

|
La comparaison des
gènes présents dans les trois domaines du
vivant est l'une des approches privilégiées
pour accéder à la connaissance de
LUCA. Ceci nécessite au
préalable l'inventaire complet des gènes
présents dans chaque domaine. Ce travail devrait
bénéficier du grand nombre de génomes
qui vont être complètement
séquencés dans les années à
venir. Un certain nombre d'entre eux sont déjà
terminés. Le colloque des Treilles se tenait en fait
à peine trois mois après la publication de la
séquence complète du génome de la
levure de boulangerie, Saccharomyces
cerevisiae , et un mois avant la publication dans
Science du premier
génome d'une archaée, Methanococcus
jannaschii . Odile
Ozier-Kalogeropoulos a donc présenté
les résultats obtenus avec l'analyse complète
de S. cerevisiae , tandis que
Nikos Kyrpides nous donnait en
avant première un aperçu de ceux obtenus en
analysant le génome de M. jannaschii .
|
|
|
Christos
Ouzounis et
Nikos Kyrpides ont
présenté un premier travail comparatif
préliminaire entre les génomes des organismes
appartenant aux trois domaines. Ce travail devrait
déboucher sur l'inventaire des protéines
communes aux trois domaines, c'est à dire
présentant des similarités de séquence
telles que l'on peut raisonnablement supposer qu'elles
dérivent toutes d'un même ancêtre commun
(en terme technique, ces protéines sont dites
homologues). Ces protéines homologues ont de fortes
chances d'avoir été présents chez
LUCA. J.
Brown a proposé l'établissement sur
Internet d'un serveur pour faciliter et coordonner ce
travail de génomique comparative, et établir
ainsi la liste de ces protéines.
|
|
D'ores
et déjà, les premier résultats
suggèrent un degré de complexité
inattendu chez LUCA. Celui-ci
aurait sans doute déjà possédé
plusieurs milliers de gènes (donc de protéines
différentes). Il semble que tous les grands
systèmes qui permettent le maintien et l'expression
du matériel génétique étaient
déjà présents, ainsi que de nombreuses
capacités métaboliques.
José Castresana
apportait de solides arguments pour avancer l'idée
iconoclaste selon laquelle les mécanismes
moléculaires de la respiration de l'oxygène
étaient déjà présents chez
LUCA, en dépit d'un
environnement en apparence dépourvu d'oxygène
! L'oxygène était-il déjà
present sur notre planète dans des niches
localisées, ou bien les mécanismes de la
respiration oxydative avaient-ils une autre fonction
à l'époque de
LUCA ?
|

|
|
|
Une
question plus générale reste
posée : la présence d'un même
gène dans les trois domaines à l'heure
actuelle est-elle toujours synonyme de sa présence
chez LUCA ? Quid des transferts
de gènes qui ont pu répandre dans l'ensemble
du monde vivant une invention apparue plus tardivement dans
l'un des trois domaines ? En particulier,
Jim Brown et
Hervé Philippe ont
montré que de nombreuses phylogénies
protéiques suggèrent le transfert d'un
très grand nombre de gènes impliqués
dans le métabolisme des bactéries vers les
eucaryotes, sans doute par l'intermédiaire des
mitochondries, organites cellulaires qui ont
évolués dans les cellules eucaryotes à
partir d'anciennes bactéries endosymbiotiques.
|

Communion autour de LUCA
|
|

|
Comprendre
l'origine et l'évolution des génomes actuels
en vue de remonter dans le temps, nécessite
également de comprendre les mécanismes de leur
évolution récente. L'analyse comparée
de génomes d'organismes proches sur le plan
évolutif est très instructive de ce point de
vue. Odile Ozier-Kalogeropoulos
a ainsi comparé une partie du génome de la
levure Kluyveromyces lactis avec celui de S.
cerevisiae , tandis que
Renaud de Rosa a comparé
le génome de deux bactéries, le colibacille
Escherichia coli , dont plus de 60% de la
séquence est connue, et le pathogène
Haemophilus influenzae , dont la séquence
a été complètement
déterminée l'année dernière. Ces
travaux mettent en évidence des différences
très importantes dans les vitesses d'évolution
d'un gène à l'autre (donc d'une
protéine à l'autre), des
phénomènes de pertes rapides (au regard de
l'évolution) et massives de certains gènes, et
enfin un grand nombre de protéines dites paralogues,
c'est à dire ayant divergé par duplication
à partir d'un gène ancestral commun.
|
|
L'évolution
rapide de la séquence de certaines protéines
rend difficile la mise en évidence de leur relation
de parenté d'un domaine à l'autre. Ceci
pourrait en partie expliquer la présence dans les
génomes complètement séquencés
de nombreux gènes codant pour des protéines
qui n'ont pas d'homologues détectables dans les deux
autres domaines. Dans certains cas, il est possible de
mettre en évidence des ressemblances d'un domaine
à l'autre au niveau de la structure des
protéines en trois dimension.
Chris Sanders a ainsi
présenté un travail d'analyse structurale qui
annonce la mise en place d'une stratégie
systématique visant à identifier le plus grand
nombre de gènes et de fonctions possibles dans les
génomes entièrement séquencés.
Le nombre de gènes ayant des homologues dans les
trois domaines (donc susceptibles d'être
déjà présents chez
LUCA) devrait ainsi augmenter
dans les années à venir.
|
|
|
|
|
Quelle racine pour l'arbre universel
?
Que
faire des gènes qui ne sont présents que dans
deux domaines ? Etaient-ils déjà
présents chez LUCA et
dans ce cas, ont-ils été perdus par la suite
dans l'un des trois domaines, ou bien sont-ils apparus dans
une branche commune à deux domaines ? Le
problème est complexe ; en effet certains
gènes ne sont présents que chez les
bactéries et les archaebactéries, d'autres
chez les archaebactéries et les eucaryotes, d'autre
encore chez les bactéries et les eucaryotes.
|

S. Miller & C. de Duve
|
|
Si
l'on met de côté le problème de
l'échantillonnage, il serait a priori plus facile de
répondre à la question posée si l'on
connaissait l'emplacement de la racine de l'arbre universel
qui relie les trois domaines entre eux. Il s'agit là
d'une question très controversée. De nombreux
auteurs situent actuellement cette racine dans la branche
des bactéries (les eucaryotes et les
archaebactéries étant dans ce cas des groupes
frères). Jim Brown a
présenté des résultats allant dans ce
sens, basés sur des arbres universels fondés
sur des protéines ayant divergé à
partir d'une duplication avant la séparation des
trois domaines.
|
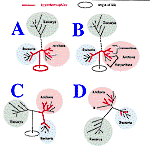
La position de LUCA et des Hyperthermophiles
|
Ce résultat pourrait expliquer la présence de
nombreux caractères de type "eucaryotes" chez les
archaées, ainsi que les phylogénies
protéines qui associent archaées et
eucaryotes. Il existe toutefois d'autres phylogénies
qui associent archaées et bactéries.
Jim Brown les interprète
en terme de transfert de gènes d'un domaine à
l'autre.
|
|

|
La
validité de ces résultats est toutefois
contestée par Patrick
Forterre et Hervé
Philippe, à partir d'études sur la
variabilité d'évolution des différentes
positions d'acides aminés d'une même
protéine. Selon eux, il ne reste plus d'information
exploitable permettant d'enraciner des arbres fondés
sur des séquences ayant divergé depuis si
longtemps. Pour Hervé
Philippe, les arbres aberrants entre protéines
des trois domaines (par exemple ceux qui ne permettent pas
de retrouver la division en trois du monde vivant) sont la
norme, et ce sont les arbres "cohérents" qui
demandent à être expliqués. Il
suggère que l'apparition de groupes
"monophylétiques" dans un arbre moléculaire
traduit un changement important dans la structure/fonction
de la molécule étudiée.
|
|
Il est
important de noter que la position de la racine de
l'arbre universel dans la branche des bactéries
favorise l'idée selon laquelle
LUCA ressemblait aux
procaryotes actuels. Néammoins, tant que la position
de la racine ne sera pas connu avec certitude, cette
question reste ouverte. En particulier, si la racine se
situe dans la branche des eucaryotes,
LUCA pourrait tout aussi bien
ressembler à une cellule de ce type !
|
|
|
|
La transition eucaryote/procaryote (ou
vice-versa ?)
Pour
Christian de Duve,
LUCA ressemblait beaucoup
à une bactérie (ou une archaée)
actuelle. Il présente un scenario astucieux qui
envisage la transition des procaryotes vers les eucaryotes
dans un milieu quasi-solide avec perte de la paroi
bactérienne suivie de l'expansion de la membrane
cytoplasmique pour permettre la phagocytose. Cette expansion
aboutissant à la formation des réseaux
membranaires intracellulaires caractéristiques des
cellules eucaryotes.
|
|
|

G. Ourisson & N. Glansdorff
|
D'autres
participants sont au contraire en faveur d'un
LUCA dont le génome
était plutôt de type eucaryote. Pour
Nicolas Glansdorff, les
opérons sont apparus tardivement, peut-être
dans une lignée commune aux archaées et aux
bactéries. Pour Rudiger
Cerff, le génome de
LUCA était
fragmenté en de nombreux chromosomes dont les
gènes contenaient des introns.
Patrick Forterre pense que
LUCA n'était ni un
procaryote, ni un eucaryote, mais un organisme de type
intermédiaire, dont les descendants ont opté
pour des stratégies adaptatives opposées :
l'adaptation vers la miniaturisation et la rapidité
maximale de réplication ayant conduit aux
procaryotes, celle visant à se nourrir d'autres
organismes par prédation ayant donné naissance
aux eucaryotes.
|
|
Pour
plusieurs participants, LUCA
n'était pas un organisme, mais une collection
d'organismes divers échangeant leurs gènes
plus ou moins sans contrainte. On retrouve à ce
propos un débat du même type que celui qui
touche au problème de l'Eve Africaine. Sans doute
aurait-il fallut associer à nos reflexions un
généticien des populations. Nous avons
regretté l'absence de Miroslav
Radman, empéché à la
dernière minute de participer à notre
colloque. Son laboratoire étudie les
mécanismes de spéciation au niveau
moléculaire, et il semble que certains d'entre eux
soient commun aux bactéries et aux eucaryotes, est-ce
à dire que les notions d'espèce et de
barrière d'espèce existaient
déjà à l'époque de
LUCA ?
|
|
|
|

|
Une
autre question essentielle reste posée, quelle
invention décisive, apparue avec
LUCA, lui a donné une
prédominance telle sur ses compétiteurs de
l'époque que seuls ses descendants peuplent
aujourd'hui notre planète ?
|
|
|
|
LUCA
était-il un hyperthermophile ?
Certains
auteurs pensent que la vie est apparue à très
haute température et que
LUCA lui-même
était un hyperthermophile (organisme vivant entre 80
et 110°C). Toutefois, Stanley
Miller et Antonio
Lazcano nous ont mis en garde contre le danger
d'extrapoler les conditions de vie supposées de
LUCA à celle des
origines. Nous avons vu en effet que
LUCA était
déjà un organisme très
sophistiqué, son apparition a donc été
précédée par une longue période
d'évolution. Stanley
Miller insiste également sur
l'instabilité de nombreux composés
prébiotiques à très haute
température. Stanley
Miller et Patrick
Forterre mettent l'accent sur la contradiction qui
existe entre l'idée d'une évolution
primordiale qui se serait entièrement produite
à haute température (de l'origine à
LUCA) et l'hypothèse du
monde à ARN, compte-tenu de l'instabilité de
cette molécule aux températures voisines du
point d'ébullition de l'eau.
Piero Cammarano présente
toutefois de nouvelles données sur l'évolution
des facteurs d'élongation (qui interviennent dans la
synthèse des protéines) en faveur de
l'ancienneté des bactéries hyperthermophiles.
Là encore, la fiabilité des analyses de
phylogénie moléculaire est au centre du
débat. Patrick Forterre
fait remarquer que les résultats obtenus avec les ARN
ribosomaux, qui sont généralement
interprêtés en faveur de l'ancienneté
des hyperthermophiles (leurs branches se signalent par une
longueur réduite), pourraient être
entachés d'erreur, étant donné leur
teneur plus élevée en résidus GC, ce
qui doit avoir pour effet de réduire artificiellement
leur vitesse d'évolution.
|
|
|
Nicolas
Glansdorff pense que la
formation des opérons a pu se produire en
réponse à l'adaptation des procaryotes aux
conditions thermophiles, tandis que
Purificacion Lopez-Garcia
présente un nouveau scénario qui envisage un
ancêtre thermophile modéré pour les
bactéries et les archaées. Ce scénario
est basé sur l'étude des enzymes qui
introduisent des superhélices dans la molécule
d'ADN (gyrase et réverse gyrase) et sur
l'hypothèse d'une géométrie
précise de cette molécule nécessaire
à son fonctionnement. Cette géométrie
aurait été initialement adaptée
à des températures aux alentours de 50
à 70°C ; la gyrase et la réverse gyrase
seraient ensuite apparues pour permettre l'adaptation des
organismes à des températures respectivement
plus basses et plus hautes. En effet, l'action de ces
enzymes permet de contrecarrer l'effet des changements de
température sur la double hélice d'ADN
(ouverture à haute température, compression
à basse température).
|
|
Que
les hyperthermophiles soient ou non plus proches de
LUCA, leur étude est un
domaine de recherche particulièrement fascinant.
Franck Robb a
présenté des expériences qui mettent en
évidence les mécanismes permettant aux
protéines des archaées hyperthermophiles de
fonctionner à des températures voisines du
point d'ébullition de l'eau. L'une des
stratégies utilisées est la formation de
réseaux d'interactions ioniques à la surface
de la protéine. Il a d'autre part
présenté les premiers résultats
montrant que les archaebactéries hyperthermophiles
sont particulièrement résistantes aux
radiations ionisantes. Ces organismes doivent
posséder des mécanismes de réparation
de l'ADN extêmement performants.
|
|
|
|
|
|
L'âge de
LUCA.
Quelques
mois avant la tenue de notre colloque, un article
publié dans la revue américaine Science avait
rajeuni LUCA. Il aurait
vécu il y a deux milliards d'années, et non
3,5 ou 4 comme on le pensait précédemment
d'après l'examen des microfossiles trouvés
dans les quelques roches sédimentaires de cette
époque encore détectables.
Hervé Philippe a soumis
les données de ce travail à une analyse
critique approfondie d'où il ressort que le chiffre
avancé de 2 milliards d'années repose sur des
données de très mauvaise qualité. Les
arbres phylogénétiques de protéines
utilisés donnaient pour la plupart des
résultats aberrants et la calibration de l'horloge
moléculaire est elle-même en question.
|

M. Fontecave & O. Ozier-Kalogeropoulos
|
|
|
|
Peut-on remonter dans le temps,
au-delà de l'époque où vivait
LUCA
?
La
complexité de
LUCA suggère qu'une
longue période (tout au moins en terme de changements
évolutif) a précédé son
émergence. Les premiers pas de la vie sur notre
planète ont été l'apparition et
l'assemblage des constituants moléculaires de la vie
jusqu'à l'apparition des premières cellules.
Le génome de ces premières cellules
était sans doute constitué par des
molécules d'ARN et non pas d'ADN. Contrairement
à l'ADN, l'ARN peut en effet jouer à la fois
le rôle d'enzyme et de matériel
génétique. Stanley
Miller et Christian de
Duve pensent que l'apparition de l'ARN a
été elle-même un événement
tardif, en effet, cette molécule ne semble pas
pouvoir être synthétiser par les
méthodes simples de la chimie prébiotique.
L'ARN aurait donc été
précédé par des molécules dont
nous ne connaitrons sans ddoute jamais la nature exacte.
|
|
|

|
Des
premières cellules à ARN jusqu'à
LUCA, le chemin a dû
être encore très long, toujours en terme de
changements évolutifs. Patrick
Forterre suggère de diviser cette
évolution en plusieurs périodes : les deux
âges du monde à ARN (avant et après
l'invention par l'ARN du système actuel de
synthèse des protéines) et le premier
âge du monde à ADN (de la première
cellule à ADN jusqu'à l'émergence de
LUCA). Comment obtenir des
informations sur ces différentes étapes ?
Selon lui ; les virus à ARN et certains virus
à ADN pourraient être des descendants
d'organismes cellulaires ayant vécu avant
LUCA. Ils n'auraient
survécu à la domination de ce dernier qu'en
parasitant ses descendants. L'étude de la biologie
moléculaire des virus pourrait donc apporter des
informations sur les étapes de l'évolution
antérieure à
LUCA.
|
|
|
Un
événement essentiel de toute cette
évolution a été l'invention de l'ADN.
Aujourd'hui, la synthèse de l'ADN dépend d'une
enzyme clef, la ribonucléotide réductase
(RNR), qui fabrique les précurseurs de cette
molécule à partir des précurseurs de
l'ARN (réduction par élimination d'un
oxygène du ribose de l'ARN pour donner du
déoxyribose). Marc
Fontecave a fait le point de nos connaissances sur
les trois classes de RNR connues à ce jour et sur
leur distribution dans le monde vivant. Il a
présenté la caractérisation par son
équipe de la première RNR
d'archaebactérie, en collaboration avec
Franck Robb. Ce travail permet
pour la première fois de montrer que les trois
classes de RNR dérivent d'une même enzyme
ancestrale. Marc Fontecave et
Franck Robb ont profité
de leur présence commune aux Treilles pour
rédiger un article rapportant cette
découverte. L'existence d'une RNR homologue dans les
trois domaines du vivant conforte l'idée selon
laquelle le génome de
LUCA était
déjà constitué d'ADN.
|
|
|
Pour
établir les portraits robots des
prédecesseurs de LUCA,
Antonio Lazcano propose
d'utiliser les protéines paralogues dont l'origine
(par duplication de gènes) est antérieure
à celle de LUCA.
Arturo Becerro a
présenté un exemple de mise en oeuvre de cette
stratégie dans l'étude de l'évolution
des gènes impliqués dans certaines voies
métaboliques. Un autre exemple d'analyse comparative
des voies métaboliques dans les trois domaines a
été présenté par
Nicolas Glansdorff. Là
encore, le problème est de distinguer duplications
ayant eu lieu avant ou après
LUCA, seules les
premières pouvant permettre de reconstituer
l'histoire des gènes antérieur à
LUCA.
Bernard Labedan et
Renaud de Rosa ont
montré en effet que les duplications récentes
sont nombreuses chez les bactéries et
Odile Ozier-Kalogeropoulos a
rapporté des résultats du même ordre
chez la levure.
|
|
|
|

P. Lopez Garcia, B. Labedan & R. deRosa
|
Bernard
Labedan a résumé
des travaux faits en collaboration avec
Monica Riley sur les
familles de protéines chez E. coli
qui suggèrent que beaucoup de protéines
ancestrales étaient déjà formées
de grands modules dont la taille et la fonction
étaient proches de celles des protéines
actuelles. Pour retrouver les homologies les plus anciennes,
il faudra sans doute en venir à une analyse des
protéines en terme modulaire et à des analyses
structurales du type de celles proposées par
Chris Sander. Le
problème sera alors de distinguer entre homologie
(structure commune héritée d'un ancêtre
commun) et analogie (structure commune acquise par
évolution convergente). En fait, tout le monde est
tombé d'accord sur la nécessité de
combiner les études génomiques (comparaison de
séquences) et les données structurales qui
peuvent être plus informatives au niveau des
fonctions, afin d'analyser l'évolution de ces
dernières, qui sont les véritables cibles de
la sélection naturelle.
|
|
|
Finalement
la question se pose : est-il possible d'extrapoler du
métabolisme actuel vers le métabolisme
ancestral ? Guy Ourisson a
présenté une tentative de ce genre dans le cas
des membranes cellulaires qu'il considère comme ayant
dû se former très tôt, peut-être
avant tout autre processus. Il a montré comment la
très grande variété des amphiphiles
membranaires actuels, souvent découverts à
partir de leurs "fossiles moléculaires" isolés
de sédiments, forme une série
évolutive. Par une analyse rétrograde, il en
déduit que les premières membranes ont pu
être formées par des phosphates de
polyprényle, qu'il a obtenus par des réactions
abiotiques - et donc peut-être prébiotiques.
|
|
|
De son
côté, Christian de
Duve remonte du métabolisme actuel jusqu'au
proto-métabolisme qui a précédé
le monde à ARN (le monde à thioesters qu'il a
popularisé dans son ouvrage Construire
une cellule). Il s'élève contre
l'idée absolutiste d'un monde à ARN
omnipotent. Selon lui, toutes les enzymes actuelles n'ont
pas été précédées par le
ribozyme correspondant, et il faut imaginer un monde de
catalyseurs peptidiques efficaces et très tôt
diversifiés. Stanley
Miller, qui avait fait le point sur l'état
actuel de la recherche dans le domaine des origines de la
vie dans sa conférence introductive, propose en
conclusion un véritable programme de recherche
expérimental pour relier la chimie prébiotique
au métabolisme contemporain et avance plusieurs
pistes nouvelles.
|

P. Cammarano, P. Lopez Garcia, A. Lazecano & A. Becerra
|
|
En
conclusion, Christian de
Duve a souligné la qualité et
l'intensité des discussions. En apparence, chacun
avait gardé son point de vue sur les questions
chaudement débattues, mais il était
évident que les arguments apportés par les uns
et les autres allaient nourrir dans les mois à venir
de nombreuses reflexions, remises en questions, ou
approfondissement des diverses hypothèses. La plupart
d'entre nous vont se retrouver pour la rédaction
d'une série d'articles à paraître dans
un numéro spécial consacré à
notre colloque par la revue internationale "Journal of
Molecular Evolution". De l'avis de plusieurs participants,
il s'agissait de l'une des meilleurs réunions
scientifiques à laquelle ils avaient eu l'occasion de
participer (le cadre des Treilles étant pour beaucoup
dans cette appréciation), et nous espèrons
tous renouveller cette expérience dans les
années à venir.
|
|
Plusieurs articles issus de ces rencontres passionnantes
( ainsi que de nouvelles collaborations engagées aux
Treilles ) ont étés publiés dans un
numéro spécial de la revue "Journal of
Molecular Evolution" Vol 49, Oct 1999.
|